Complexitate adăugată –
Schimbările majore sub capotă îi permit lui AlphaFold să gestioneze complexele proteine-ADN și multe altele.
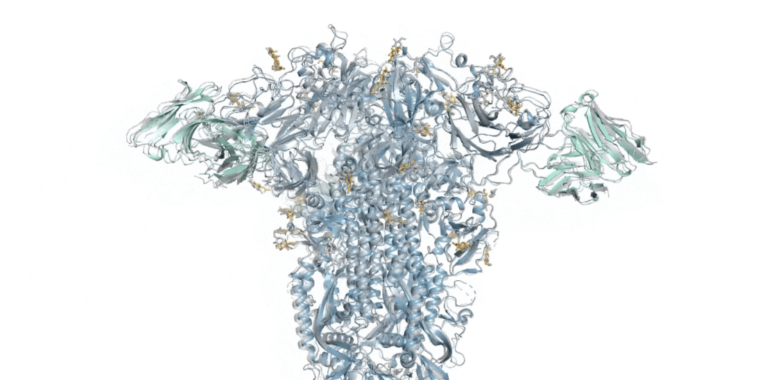
Mărește / Predicția structurii unui coronavirus Spike proteină dintr-un virus care provoacă răceala comună.
Google DeepMind
Majoritatea activităților care se desfășoară în interiorul celulelor – activitățile care ne țin să trăim, să respirăm, să gândim animalele – sunt gestionate de proteine. Acestea permit celulelor să comunice între ele, să conducă metabolismul de bază al celulei și să transforme informațiile stocate în ADN în și mai multe proteine. Și toate acestea depind de capacitatea șirului de aminoacizi a proteinei de a se plia într-o formă tridimensională complicată, dar specifică, care îi permite să funcționeze.
Până în acest deceniu, înțelegerea că forma 3D a însemnat purificarea proteinei și supunerea acesteia unui proces care necesită timp și muncă pentru a determina structura acesteia. Dar asta s-a schimbat odată cu munca lui DeepMind, una dintre diviziile AI ale Google, care a lansat Alpha Fold în 2021și a efort academic similar la scurt timp după aceea. Software-ul nu a fost perfect; s-a luptat cu proteine mai mari și nu a oferit soluții de mare încredere pentru fiecare proteină. Dar multe dintre predicțiile sale s-au dovedit a fi remarcabil de precise.
Chiar și așa, aceste structuri au spus doar jumătate din poveste. Pentru a funcționa, aproape fiecare proteină trebuie să interacționeze cu altceva – alte proteine, ADN, substanțe chimice, membrane și multe altele. Și, în timp ce versiunea inițială a AlphaFold ar putea face față unor interacțiuni proteină-proteină, restul au rămas cutii negre. Astăzi, DeepMind anunță disponibilitatea versiunii 3 a AlphaFold, care a văzut părți ale motorului său de bază fie puternic modificate, fie înlocuite în întregime. Datorită acestor modificări, software-ul gestionează acum diverse interacțiuni și modificări suplimentare de proteine.
Schimbarea pieselor
AlphaFold original s-a bazat pe două funcții software de bază. Una dintre acestea a luat în considerare limitele evolutive ale unei proteine. Privind aceeași proteină în mai multe specii, puteți obține o idee pentru care părți sunt întotdeauna aceleași și, prin urmare, probabil să fie esențial pentru funcția sa. Această centralitate implică faptul că ei sunt întotdeauna probabil în aceeași locație și orientare în structura proteinei. Pentru a face acest lucru, AlphaFold original a găsit cât mai multe versiuni ale unei proteine și și-a aliniat secvențele pentru a căuta porțiunile care au prezentat puține variații.
A face acest lucru, totuși, este costisitor din punct de vedere computațional, deoarece cu cât aliniați mai multe proteine, cu atât trebuie să rezolvați mai multe constrângeri. În noua versiune, echipa AlphaFold a identificat în continuare mai multe proteine înrudite, dar a trecut la aliniamente care realizează în mare măsură, folosind perechi de secvențe de proteine din setul de cele înrudite. Acest lucru probabil nu este la fel de bogat în informații ca o multi-aliniere, dar este mult mai eficient din punct de vedere computațional, iar informațiile pierdute nu par a fi esențiale pentru a descoperi structurile proteinelor.
Folosind aceste alinieri, un modul software separat a descoperit relațiile spațiale dintre perechile de aminoacizi din proteina țintă. Aceste relații au fost apoi traduse în coordonate spațiale pentru fiecare atom prin cod care a ținut cont de unele dintre proprietățile fizice ale aminoacizilor, cum ar fi porțiunile unui aminoacid s-ar putea roti în raport cu altele etc.
În AlphaFold 3, predicția pozițiilor atomice este gestionată de un modul de difuzie, care este antrenat oferindu-i atât o structură cunoscută, cât și versiuni ale acelei structuri în care s-a adăugat zgomot (sub formă de deplasare a pozițiilor unor atomi). Acest lucru permite modulului de difuzie să ia locațiile inexacte descrise de poziții relative și să le transforme în predicții exacte ale locației fiecărui atom din proteină. Nu trebuie să i se spună proprietățile fizice ale aminoacizilor, deoarece își poate da seama ce fac ei în mod normal uitându-se la suficiente structuri.
(DeepMind a trebuit să se antreneze pe două niveluri diferite de zgomot pentru a face modulul de difuzie să funcționeze: unul în care locațiile atomilor au fost deplasate în timp ce structura generală a fost lăsată intactă și al doilea în care zgomotul implica schimbarea structurii la scară largă a proteine, afectând astfel localizarea multor atomi.)
În timpul antrenamentului, echipa a descoperit că a fost nevoie de aproximativ 20.000 de instanțe de structuri de proteine pentru ca AlphaFold 3 să obțină corect aproximativ 97% dintr-un set de structuri de testare. În 60.000 de cazuri, a început să obțină interfețele proteine-proteine corecte și la acea frecvență. Și, în mod critic, a început să primească și proteinele complexate cu alte molecule.