Mecanismele enzimatice pot fi complexe, iar a le face să funcționeze este dificil.
Enzimele sunt catalizatori uimitori. Aceste proteine nu sunt făcute din altceva decât o mână de elemente care nuanțează pe pământ și promovează o gamă vastă de reacții, transformă energia chimică în mișcare fizică și acționează cu o specificitate remarcabilă. În multe cazuri, ne-am străduit să găsim catalizatori non-enzimatici care pot conduce unele din aceleași reacții chimice.
Din păcate, nu există o enzimă pentru multe reacții pe care le -am dori cu mult să catalizăm – lucruri precum digerarea materialelor plastice sau încorporarea dioxidului de carbon în molecule mai complexe. Am avut câteva succese folosind evoluția direcționată pentru a crea variații utile ale enzimelor existente, dar eforturile de lărgire a domeniului de aplicare a ceea ce enzimele pot face au fost limitate.
Cu toate acestea, odată cu apariția proiectării proteice conduse de AI, acum putem proiecta lucruri care sunt spre deosebire de orice este găsit în natură. O nouă lucrare descrie astăzi un succes în realizarea unei enzime noi, cu potențialul de a digera materialele plastice. Dar arată, de asemenea, cum chiar și o simplă enzimă poate avea un mecanism extrem de complex – și unul greu de abordat, chiar și cu cele mai recente instrumente AI.
Finisatori esteri
Reacția la care a lucrat echipa de cercetare (implicând unii dintre aceiași oameni care Inhibitori de venin de șarpe proiectat) este defalcarea a ceea ce se numește obligațiune ester. Legăturile esterului sunt formate prin conectarea a două lanțuri de atomi de carbon de către un atom de oxigen, unul dintre carbonii flancați fiind legat de un al doilea oxigen. Acestea pot fi rupte prin adăugarea unei molecule de apă, care lasă un lanț de carbon legat de un grup de alcool (COH) și celălalt un acid organic (COOH).
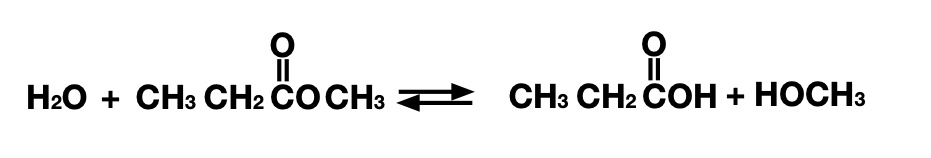
Legăturile esterului pot fi rupte prin adăugarea unei molecule de apă. Credit: John Timmer
Aceste legături apar în diferite biomolecule, astfel încât există multe enzime care le pot manipula. Dar, dincolo de biologie, acestea apar și într -o serie de polimeri din plastic pe care îi folosim la scară largă – Polyester și -a primit numele din cauza câte cazuri ale legăturii chimice apar în ea. Așadar, există o mulțime de valoare potențială în a putea descompune obligațiunile Ester. Și avem o mulțime de exemple din biologie pentru a ne arăta cum se face.
În acest caz, însă, biologia ne spune că ceea ce pare o reacție chimică simplă poate fi incredibil de complexă. Ca parte a unei serii de reacții care rup esterul în două părți, una dintre părți ajunge să fie legată chimic de un aminoacid din enzimă în sine. Această legătură trebuie să fie ruptă din nou de una dintre celelalte reacții, sau enzima devine inactivată.
Pentru a obține toate aceste reacții la lucru, enzimele au toate un aminoacid cheie care este într -un punct critic în raport cu pH -ul tipic al lucrurilor vii. Asta înseamnă că poate trage un proton din apa care o înconjoară și îl poate dona unul dintre aminoacizii din proteină. În alte puncte, în schimb, scoate un proton de pe un aminoacid, pierzându -l în una dintre părțile esterului. În general, ruperea simplă a unei legături chimice are cel puțin patru etape distincte și necesită mai mulți aminoacizi care să fie poziționați în situsul activ al enzimei cu precizie atomică.
Este foarte ușor să obțineți un instrument AI pentru a proiecta o proteină care are configurația potrivită pentru a face unul dintre acești pași. A -l ciclul prin toate cele patru este o chestiune diferită în întregime.
Aveți nevoie de mai mult AI
Cercetătorii au început folosind instrumentele standard pe care le -au dezvoltat pentru a gestiona proiectarea proteinelor, inclusiv un instrument AI numit RFDiffusion, care folosește o sămânță aleatorie pentru a genera o varietate de fundaluri proteice. În acest caz, cercetătorii au cerut RFDiffusion să se potrivească cu pozițiile medii ale aminoacizilor într-o familie de enzime care se distrug ester. Rezultatele au fost alimentate într -o altă rețea neuronală, care a ales aminoacizii, astfel încât să formeze un buzunar care să țină un ester care să se descompună într -o moleculă fluorescentă, astfel încât să poată urmări activitatea enzimei folosind strălucirea acesteia.
Din cele 129 de proteine proiectate de acest software, doar două dintre ele au dus la orice fluorescență. Așa că echipa a decis că au nevoie de încă o AI. Numit Placer, software -ul a fost instruit luând toate structurile cunoscute de proteine fixate pe molecule mici și randomizând o parte din structura lor, forțând AI -ul să învețe cum să schimbe lucrurile într -o stare funcțională (făcând -o un AI generativ). Speranța era că Placer va fi instruit pentru a surprinde unele dintre detaliile structurale care permit enzimelor să adopte mai multe configurații specifice pe parcursul reacției pe care o catalizează.
Și a funcționat. Repetarea aceluiași proces cu o etapă de screening a plăcuței adăugată a sporit numărul de enzime cu activitate catalitică de peste trei ori.
Din păcate, toate aceste enzime s -au oprit după o singură reacție. Se dovedește că au fost mult mai buni la clivarea esterului, dar au lăsat o parte din ea legată chimic de enzimă. Cu alte cuvinte, enzimele au acționat ca o parte a reacției, nu ca un catalizator. Așadar, cercetătorii au început să folosească Placer pentru a ecraniza structurile care ar putea adopta o stare intermediară cheie a reacției. Aceasta a produs o rată mult mai mare de enzime reactive (18 la sută dintre ei au clivat obligațiunea esterului), iar două – numite „Super” și „Win” – ar putea parcurge efectiv mai multe runde de reacții. Echipa făcuse în sfârșit o enzimă.
Prin adăugarea de runde suplimentare alternând între sugestiile structurii folosind RFDiffusion și screening folosind Placer, echipa a văzut că frecvența enzimelor funcționale crește și, în cele din urmă, a proiectat una care a avut o activitate similară cu unele produse de lucrurile vii. De asemenea, au arătat că ar putea folosi același proces pentru a proiecta o esterază capabilă să digere legăturile din PET, un plastic comun.
Dacă sună ca multă muncă, a fost clar – proiectarea enzimelor, în special a celor în care știm despre enzime similare în lucrurile vii, vor rămâne o provocare serioasă. Dar cel puțin o mare parte din acesta se poate face pe computere, mai degrabă decât să solicite cuiva să comande ADN -ul care codifică enzima, să obțină bacterii să o facă și să depună screening pentru activitate. Și în ciuda procesului care implică referințe la enzimele cunoscute, cele proiectate nu au împărtășit o mulțime de secvențe în comun cu ele. Acest lucru sugerează că ar trebui să fie adăugate flexibilitate dacă dorim să proiectăm unul care să reacționeze cu esteri pe care lucrurile vii nu s -au întâlnit niciodată.
Sunt curios despre ce s -ar putea întâmpla dacă proiectăm o enzimă care este esențială pentru supraviețuire, o punem în bacterii și apoi îi permitem să evolueze o perioadă. Bănuiesc că viața ar putea găsi modalități de îmbunătățire a celor mai bune modele ale noastre.
Știință, 2024. DOI: 10.1126/Science.adu2454 (Despre Dois)
John este editorul științific al ARS Technica. Are un licențiat în arte în biochimie de la Universitatea Columbia și un doctorat. în biologie moleculară și celulară de la Universitatea din California, Berkeley. Când se desparte fizic de tastatura sa, el tinde să caute o bicicletă sau o locație pitorească pentru comunicarea cu cizmele sale de drumeție.

