Obținerea CRISPRy —
Unele sunt legate de enzimele de tăiere a ADN-ului. Altele sunt un mister complet.
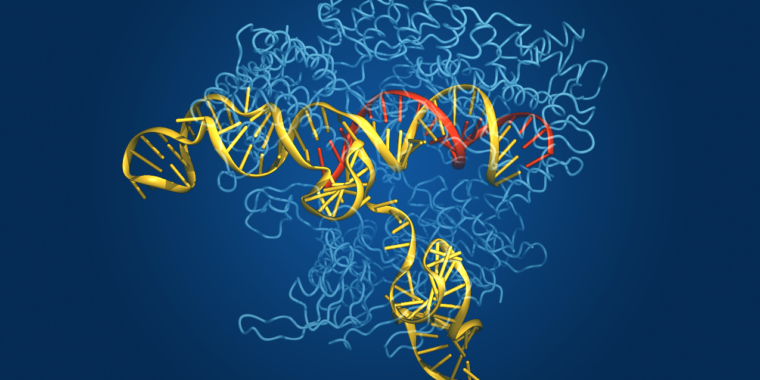
Mărește / Structura proteică a CAS, prezentată cu acizii nucleici legați.
CRISPR—Clustered Regularly Interspaced Short Palindromic Repeats—este răspunsul lumii microbiene la imunitatea adaptivă. Bacteriile nu generează anticorpi atunci când sunt invadate de un agent patogen și apoi țin acești anticorpi în așteptare în cazul în care întâlnesc din nou același agent patogen, așa cum facem noi. În schimb, ei încorporează o parte din ADN-ul patogenului în propriul lor genom și îl leagă de o enzimă care o poate folosi pentru a recunoaște acea secvență de ADN patogen și o taie în bucăți dacă agentul patogen apare din nou.
Enzima care face tăierea se numește Cas, pentru CRISPR asociat. Deși sistemul CRISPR-Cas a evoluat ca mecanism de apărare bacteriană, a fost valorificat și adaptat de cercetători ca un instrument puternic pentru manipularea genetică în studiile de laborator. De asemenea, a demonstrat utilizări în agricultură, iar prima terapie bazată pe CRISPR tocmai a fost aprobată în Marea Britanie pentru a trata boala sicletică și beta-talasemia dependentă de transfuzii.
Acum, cercetătorii au dezvoltat o nouă modalitate de a căuta în genomi sisteme asemănătoare CRISPR-Cas. Și au descoperit că este posibil să avem o mulțime de instrumente suplimentare cu care să lucrăm.
Modificarea ADN-ului
Până în prezent, șase tipuri de sisteme CRISPR-Cas au fost identificate în diverși microbi. Deși diferă în detaliu, toate au același atractiv: furnizează proteine la o anumită secvență de material genetic cu un grad de specificitate care până acum a fost dificil din punct de vedere tehnic, costisitor și consumator de timp. Orice secvență de ADN de interes poate fi programată în sistem și țintită.
Sistemele native găsite în microbi aduc de obicei o nuclează – o enzimă de scindare a ADN-ului – la secvență, pentru a tăia materialul genetic al unui agent patogen. Această capacitate de a tăia orice secvență de ADN aleasă poate fi utilizată pentru editarea genelor; în tandem cu alte enzime și/sau secvențe de ADN, poate fi folosit pentru a introduce sau șterge secvențe scurte suplimentare, corectând genele mutante. Unele sisteme CRISPR-Cas scindează molecule specifice de ARN în loc de ADN. Acestea pot fi folosite pentru a elimina ARN-ul patogen, cum ar fi genomul unor virusuri, în felul în care sunt eliminați în bacteriile lor native. Acest lucru poate fi folosit și pentru a salva defectele procesării ARN.
Dar există o mulțime de modalități suplimentare de a modifica acizii nucleici care ar putea fi utile. Și este o întrebare deschisă dacă enzimele care efectuează modificări suplimentare au evoluat. Așa că, unii cercetători au decis să le caute.
Cercetătorii de la MIT au dezvoltat un nou instrument pentru a detecta matrice variabile CRISPR și l-au aplicat la perechi de baze de 8,8 tera (1012) de informații genomice procariote. Multe dintre sistemele pe care le-au găsit sunt rare și au apărut în setul de date doar în ultimii 10 ani, evidențiind cât de important este să continuăm să adăugați mostre de mediu care anterior erau greu de obținut în aceste depozite de date.
Noul instrument a fost necesar deoarece bazele de date cu secvențe de proteine și acid nucleic se extind într-un ritm ridicol, așa că tehnicile de analiză a tuturor acestor date trebuie să țină pasul. Unii algoritmi care sunt utilizați pentru a le analiza încearcă să compare fiecare secvență cu oricare alta, ceea ce este evident de nesuportat atunci când avem de-a face cu miliarde de gene. Alții se bazează pe grupare, dar aceștia găsesc doar gene care sunt foarte asemănătoare, așa că nu pot face cu adevărat lumină asupra relațiilor evolutive dintre proteinele înrudite la distanță. Dar gruparea rapidă bazată pe hashtag-uri („flash cluster”) funcționează prin gruparea miliardelor de proteine în grupuri mai puține și mai mari de secvențe care diferă ușor pentru a identifica rude noi, rare.
Căutarea folosind FLSHclust a scos cu succes 188 de noi sisteme CRISPR-Cas.
O mulțime de CRISPyness
Câteva teme au apărut din lucrare. Una este că unele dintre sistemele CRISPR nou identificate folosesc enzime Cas cu domenii nemaivăzute până acum sau par a fi fuziuni cu gene cunoscute. Oamenii de știință au caracterizat în continuare unele dintre acestea și au descoperit că una este mai specifică decât enzimele CRISPR utilizate în prezent, iar alta care reduce ARN-ul pe care o propun este suficient de distinctă din punct de vedere structural pentru a cuprinde un al șaptelea tip complet nou de sistem CRISPR-Cas.
Un corolar al acestei teme este legarea enzimelor cu diferite funcționalități, nu doar nucleaze (enzime care taie ADN-ul și ARN-ul), cu matrice CRISPR. Oamenii de știință au valorificat capacitatea remarcabilă de direcționare a genelor a CRISPR, legându-l de alte tipuri de enzime și molecule, cum ar fi coloranții fluorescenți. Dar evoluția, evident, a ajuns prima.
Ca un exemplu, FLSHclust a identificat ceva numit transposază asociată cu două tipuri diferite de sisteme CRISPR. O transpozază este o enzimă care ajută o anumită porțiune de ADN să sară în altă parte a genomului. Transpunerea ghidată de ARN CRISPR a fost văzută înainte, dar acesta este un alt exemplu al acesteia. O serie întreagă de proteine cu funcții diferite, cum ar fi proteinele cu domenii transmembranare și molecule de semnalizare, au fost găsite legate de matricele CRISPR, evidențiind natura mix-n-match a evoluției acestor sisteme. Ei au găsit chiar și o proteină exprimată de un virus care se leagă de matricele CRISPR și le face inactive – în esență, virusul inactivează sistemul CRISPR care a evoluat pentru a proteja împotriva virușilor.
Cercetătorii nu numai că au găsit noi proteine asociate cu matricele CRISPR, dar au găsit și alte matrice repetate interspațiate în mod regulat care nu au fost asociate cu nicio enzimă cas – similare cu CRISPR, dar nu CRISPR. Ei nu sunt siguri care ar putea fi funcționalitatea acestor sisteme ghidate de ARN, dar speculează că sunt implicați în apărare la fel ca CRISPR.
Autorii și-au propus să găsească „un catalog de proteine ghidate de ARN care să ne extindă înțelegerea biologiei și evoluției acestor sisteme și să ofere un punct de plecare pentru dezvoltarea de noi biotehnologii.” Se pare că și-au atins obiectivul: „Rezultatele de această lucrare dezvăluie o flexibilitate organizațională și funcțională fără precedent și modularitatea sistemelor CRISPR”, scriu ei. Ei continuă la concluzia: “Acesta reprezintă doar o mică parte din sistemele descoperite, dar luminează vastitatea și potențialul neexploatat al biodiversității Pământului și candidații rămași vor servi drept resursă pentru explorarea viitoare.”
Articolul DOI: 10.1126/science.adi1910